近日,控制与计算机工程学院杨静教授课题组依托科技部“生物与信息融合(BT与IT融合)”重点专项《基于非传统分子比特 DNA存储的新型信息安全技术研究》的支持,在国际顶级期刊Nature Communications在线发表了题为” DNA Computing Function Switching by Programming Base Stacking Interactions with Minimal Molecular Architecture Changes”的重要研究论文。我院博士生张勇鹏为第一作者,杨静教授为第一通讯作者。
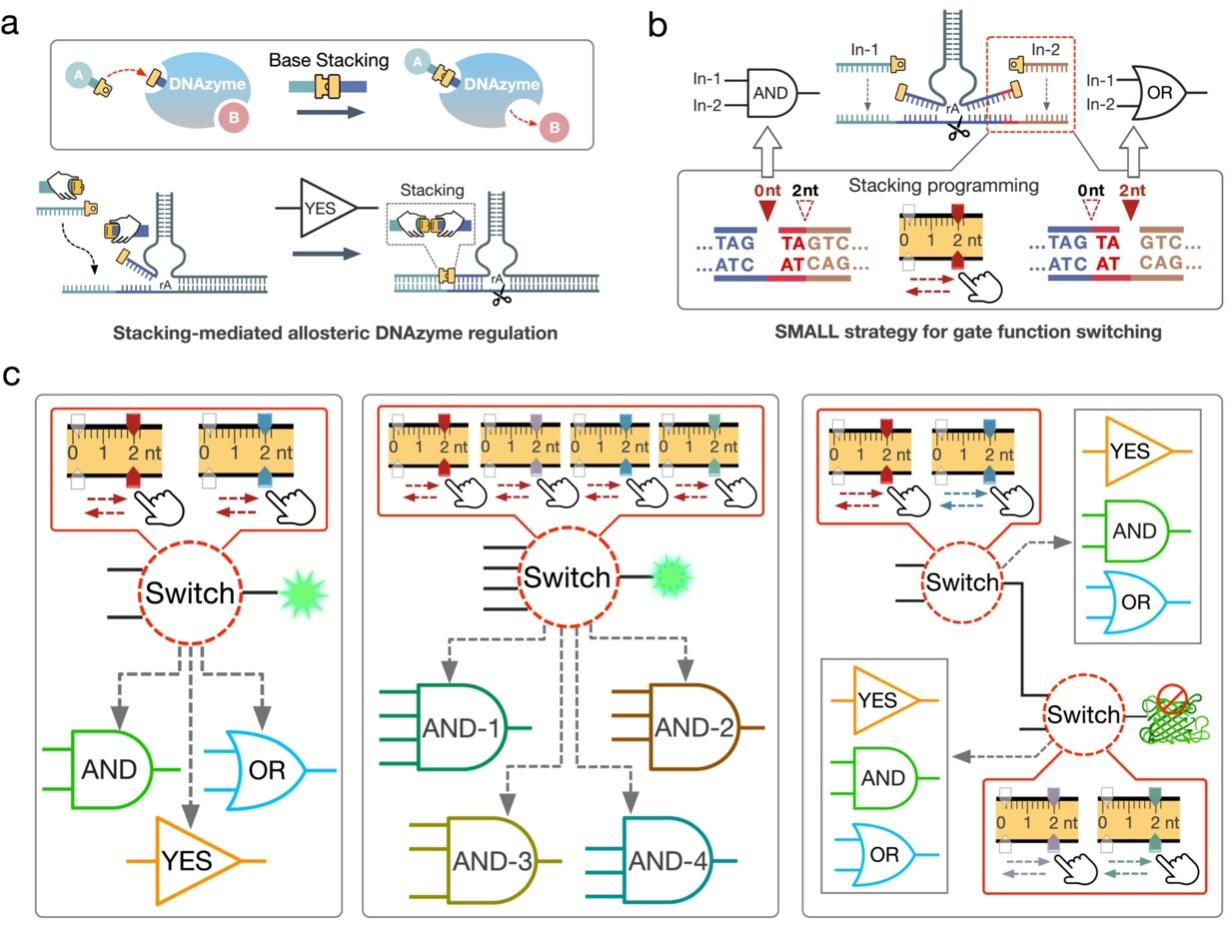
图1 基于DNA碱基堆叠力介导的变构策略(SMALL)的功能切换网络示意图
本项研究聚焦于纳米分子计算领域的功能切换问题,首次构建了基于DNA分子范德华力调控的超灵敏功能切换机制。研究团队提出一种基于DNA碱基堆积力的变构策略(SMALL),通过极小的分子结构调控即可实现DNA纳米分子计算功能的高效切换。利用该策略,团队在DNA分子计算网络中成功实现了多达20种不同的分子逻辑功能切换。同时,通过系统性实验验证,该功能切换平台在癌细胞系中可实现多达84种基因调控模式,充分展示了其在纳米分子计算与分子传感方面的应用潜力。该策略为拓展纳米分子计算电路的调控能力提供了简洁高效的解决方案。
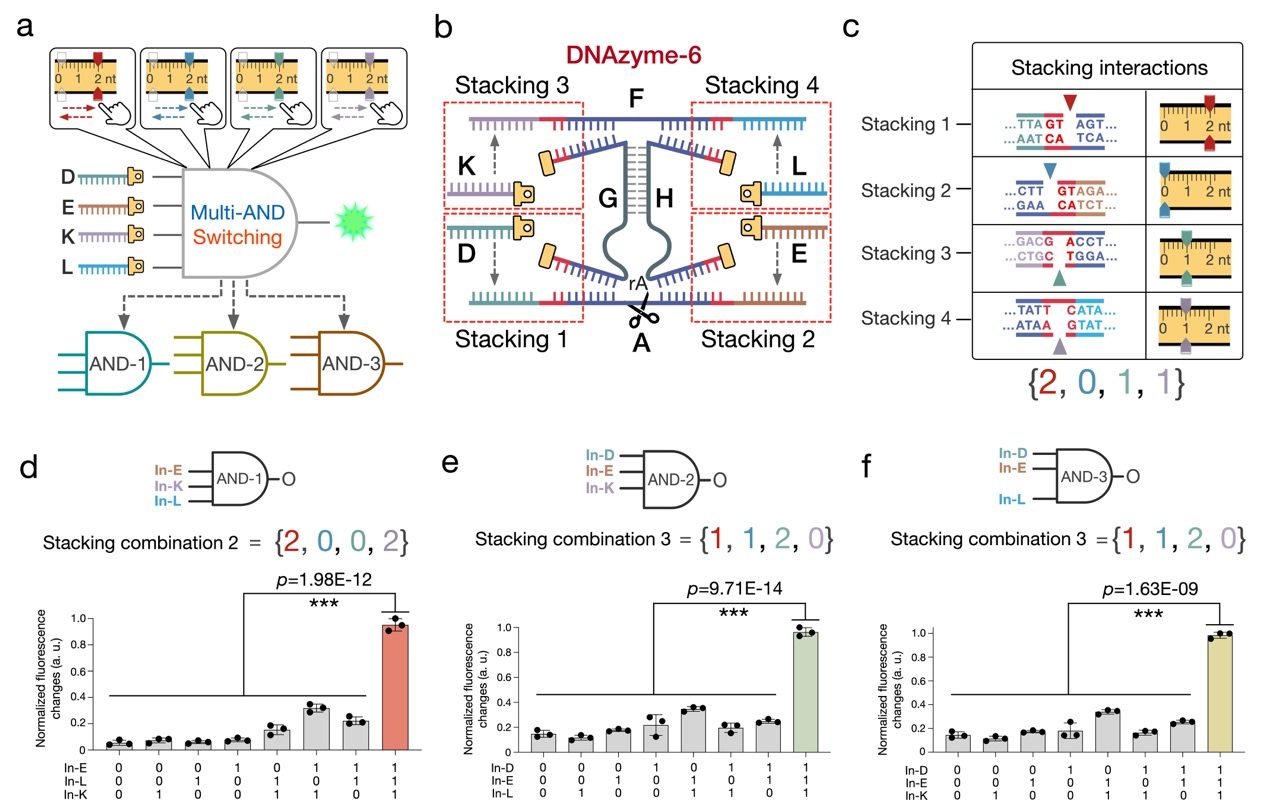
图2 多输入SMALL 分子电路逻辑门的功能切换示意图
值得注意的是,本项研究提出的SMALL策略,在操作效率与设计理念上展现出显著优势。长期以来,DNA纳米分子计算功能的切换主要依赖基于Toehold位点介导的链置换技术,该方法通常需要对DNA序列进行大规模、复杂的重排设计,步骤繁琐且容错性较低。相比之下,SMALL策略通过一种创新的局部结构调控机制,仅需精准改动1至4个关键核苷酸,即可高效、可靠地实现计算功能的切换。该策略显著简化了DNA纳米分子计算功能切换的操作流程,同时提升了设计的灵活性,为构建更复杂、更动态的DNA计算系统提供了新途径。
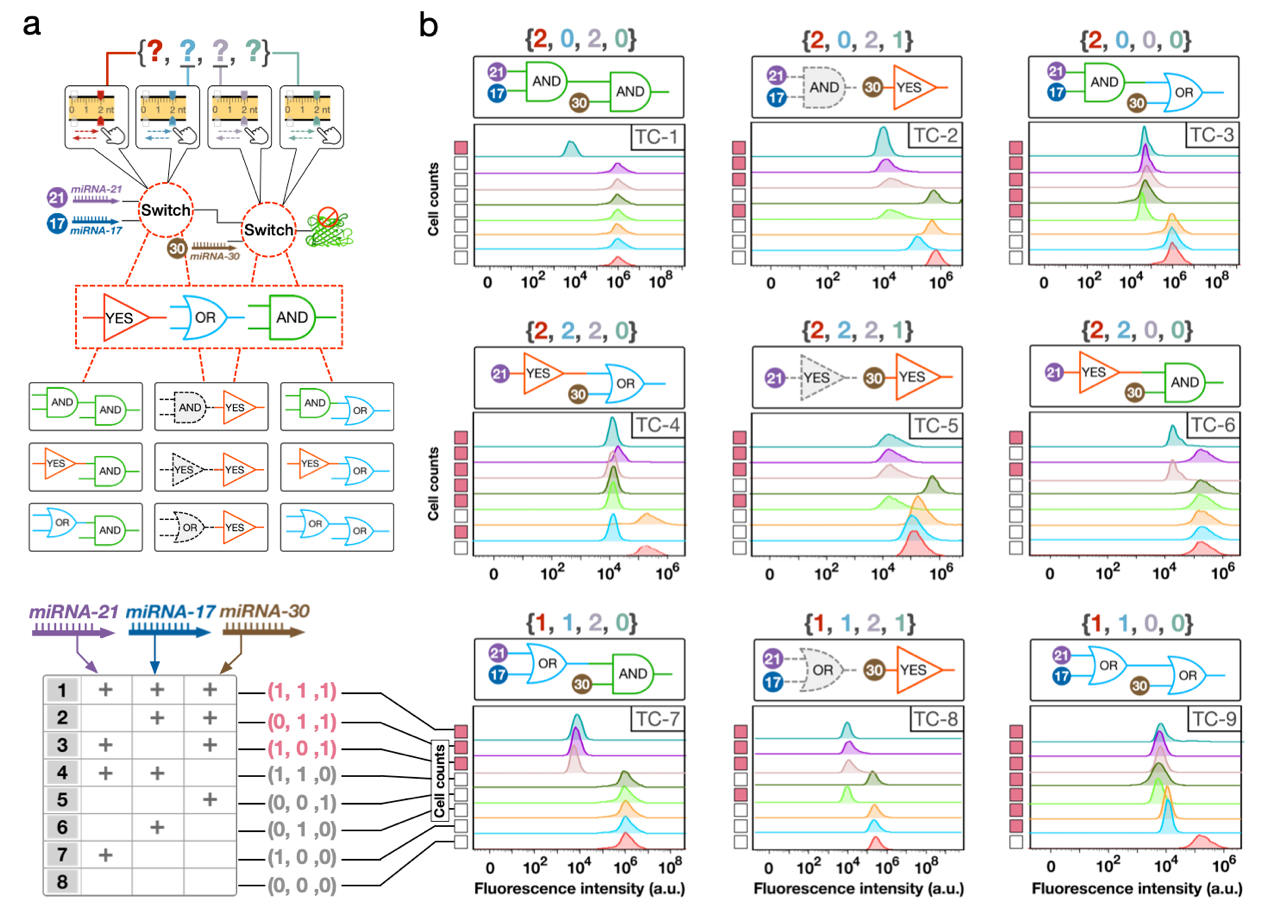
图3 级联SMALL分子逻辑网络功能切换的多模态示意图
总之,本研究提出的基于DNA碱基堆积力的变构策略(SMALL),其核心突破在于将DNA分子纳米计算功能的切换难度降低至前所未有的水平。研究结果表明,仅需对极少量核苷酸进行微调,即可实现以往依赖大规模序列重排才能完成的功能转换。该技术有望显著降低DNA分子纳米计算电路重构的复杂度,为开发下一代可编程纳米分子系统、智能纳米传感工具以及活细胞内的精准基因调控应用提供关键支撑。
杨静教授研究团队聚焦纳米分子存储与计算的前沿领域,构建了以DNA数据存储、DNA分子计算电路和新型纳米器件为核心的研究体系。团队面向国家重大战略需求,围绕前瞻性分子信息处理技术全链条—“DNA存储-计算-操控读取”开展系统研究,重点突破新型分子信息比特的并行写入技术、物理微空间中数据结构基本单元的设计与构建,以及智能化DNA分子计算电路的开发等关键科学问题,在大规模DNA分子信息存储、纳米分子信号网络调控、复杂分子信号数据挖掘与分析等方面取得了重要突破。近年来,团队依托生物与信息融合(BT与IT融合)国家重点研发计划,在国际顶级期刊Nature, Nature Communications、Science Advances等发表系列高水平学术成果。

初审:杨静
复审:陈希
审核:王韶华